
Resistencia a antibióticos: un problema local con consecuencias globales
El descubrimiento de la Penicilina, de la mano de Alexander Fleming en 1928, marcó un antes y un después en la medicina. Este nuevo fármaco salvó miles de vidas de soldados durante la Segunda Guerra Mundial, que de otra manera hubiesen muerto de simples infecciones bacterianas en heridas menores. Desde ese momento, se han descubierto un gran arsenal de nuevos antibióticos, moléculas naturales que se han modificado químicamente para ser más activas o sintéticas, siendo todos ellos la base de la medicina actual. Además de curar infecciones, sin ellos no podríamos enfrentarnos a operaciones quirúrgicas sencillas, utilizar quimioterapia en pacientes con cáncer o realizar trasplantes de órganos (Figura 1).

En las últimas décadas venimos asistiendo a un alarmante incremento en la aparición y propagación de infecciones causadas por bacterias resistentes a antibióticos. Este problema está cada vez más presente en los medios de comunicación nacionales, que cada semana muestran noticias relacionadas con este grave problema y sus consecuencias. Sin embargo, sigue siendo desconocido para gran parte de nuestra sociedad. Ésta es una de las amenazas más severas a las que nos enfrentamos en salud pública, y es uno de los mayores retos para la medicina moderna.
Según los datos de los organismos sanitarios europeos, nada más y nada menos que 8 personas al día mueren por causa de una infección bacteriana resistente a los antibióticos en España. Esto son 3.000 personas al año y 33.000 Europa. Las previsiones para 2050 no son muy optimistas. Se estima que este valor supere las 40.000 muertes en España, multiplicando por 13 el valor actual en tan sólo 35 años. En Europa serían un total de 390.000 muertes anuales. La Organización Mundial de la Salud, ha alertado que estas muertes superarán a las producidas por cáncer en la población mundial. Todo esto, si las previsiones se cumplen y los antibióticos dejan de funcionar correctamente. Si nos fijamos en términos económicos, este problema nos supone a los españoles un gasto de 150 M de euros anuales, aumentando el gasto por paciente entre 5,000 a 15,000 E. En 2050 la estimación sitúa este coste en 80 billones de euros para toda la Unión Europea.
¿Pero a qué se debe este aumento en las tasas de resistencia a antibióticos? Aunque existen diversos factores, quizás el más notable es el uso inapropiado e indiscriminado de estos fármacos en medicina humana y veterinaria. Podemos resumir en seis los problemas a los que nos enfrentamos. En primer lugar, la utilización inadecuada de los antibióticos, tanto por una prescripción errónea como por un mal uso por parte del paciente. Debemos tener también en cuenta el uso intensivo de estos fármacos en hospitales. Éstos son lugares donde la concentración de personas que están recibiendo este tipo de tratamiento es muy alta, por lo que son refugios para las bacterias resistentes. Esto nos obliga a optimizar y reducir este uso, implementar las medidas de control y de higiene óptimas para el manejo de pacientes, así como a mejorar las técnicas de identificación y vigilancia de estas bacterias peligrosas. Seguido de este uso se encuentra la utilización masiva de antibióticos en cría de animales destinados a consumo humano. Aunque actualmente está limitado al tratamiento terapéutico, éstos han sido en el pasado, o son clandestinamente hoy en día, alimentados o tratados con toneladas de antibióticos. Otro de los problemas principales es la falta de nuevos fármacos de este tipo. Desde el descubrimiento de la penicilina, y especialmente durante los años 60-70, hubo un incremento notable de nuevas moléculas. Sin embargo, en las últimas dos décadas apenas se han descubierto una docena de nuevos tipos de antibióticos, por lo que las empresas farmacéuticas han dejado de investigar en ellos. La mayoría de los que utilizamos a diario están dejando de funcionar y nos vemos en la necesidad de aumentar las dosis o volver a utilizar aquellos descartados por su alta toxicidad. El quinto problema, y aunque parezca alejado del problema del que les hablamos, se encuentra el cambio climático. Los eventos climáticos extremos causarán un aumento de las enfermedades infecciosas y por tanto, una necesidad de utilizar estos fármacos de manera masiva en las zonas afectadas. Y finalmente, el sexto problema, es que las bacterias son viajeras. Viajan con nosotros, con los alimentos y con los animales, aumentando así, el riesgo de diseminación entre países y continentes distintos. Las infecciones ya no son locales, ahora son globales.
Por lo tanto, la resistencia a antibióticos es un problema global, que no sólo afecta a la salud humana, sino también a sanidad animal, ganadería, agricultura, medio ambiente, comercio y por tanto, a la economía mundial. Dada esta característica global del problema, el enfoque que se propone desde diversos organismos internacionales es un enfoque denominado “One Health”, una única salud, donde se traten cada uno de estos factores, no de manera aislada, sino coordinada, en la búsqueda de soluciones transversales.
Ante este panorama desolador, la Unión Europea ha establecido como una de las líneas prioritarias la lucha contra la resistencia a antibióticos, publicando el Plan Director de Acción sobre Resistencias en 2011, y estimulando la creación de los planes nacionales, presentes en 13 países. Nuestro país aprobó el Plan Nacional frente a la Resistencia a Antibióticos (PRAN) en 2014. (Figura 2).

Este plan involucra a todas las Comunidades Autónomas, 8 ministerios, 70 sociedades científicas y más de 300 expertos. Está vertebrado en seis áreas de trabajo que incluyen, la vigilancia en el consumo de antibióticos y aparición de resistencias; el control, mediante la elaboración de guías terapéuticas y prescripción responsable; la prevención, mediante programas de formación tanto para profesionales sanitarios del ámbito hospitalario como para veterinarios; la investigación, con el fin de desarrollar una estrategia común en desarrollo de nuevos antibióticos, así como impulsar el conocimiento básico sobre el problema; la formación, para concienciar desde las etapas más tempranas de la educación; y la comunicación, con el fin de sensibilizar a la población general.
Entre las iniciativas de comunicación propuestas por el Plan Nacional se fomenta la participación de los investigadores, personal sanitario y personal docente en los premios PRAN. Este año se ha celebrado su segunda edición, y se han otorgado un total de 5 premios, durante la celebración del día europeo para el uso prudente de antibióticos el pasado 18 de noviembre. Podemos contar con orgullo que uno de los artículos en los que hemos trabajado durante los últimos años ha sido galardonado en la categoría de mejor publicación científica sobre antibióticos y resistencia. Este trabajo se titula “Secuenciación de genoma completo, tipificación molecular y virulencia en vivo de aislados de Escherichia coli productora de la carbapenemasa OXA-48”. Aborda desde un punto de vista multidisciplinar el grave problema de la resistencia a carbapenémicos, uno de los antibióticos de última generación disponibles para el tratamiento de infecciones multirresistentes.
El equipo de este trabajo está formado por biólogos, veterinarios y bioinformáticos, y lo componemos la Dra. Rosario Rodicio (Universidad de Oviedo), Dr. Jorge Blanco y Dra. Azucena Mora (U. Santiago de Compostela, Campus de Lugo), Dr. Fernando de la Cruz (U. de Cantabria), Dra. Vanesa García (actualmente en la U. Copenhague), Dr. Javier Fernández (Hospital Universitario Central de Asturias) y Dra. María de Toro (Centro de Investigación Biomédica de La Rioja). (Figura 3).

Nuestro estudio comenzó cuando observamos en el Hospital Universitario Central de Asturias un repunte en el número de pacientes que mostraban infecciones bacterianas causadas por cepas resistentes a antibióticos carbapenémicos durante los años 2012 a 2015. Entre el conjunto de bacterias que producían estas infecciones seleccionamos para nuestro estudio 7 muestras de Escherichia coli (E. coli), resistentes no solo a este antibiótico, sino también y al menos, a otras dos familias de antibióticos. Por lo tanto, eran consideradas bacterias multirresistentes. Éstas procedían de pacientes que tenían heridas quirúrgicas o infecciones de orina y que habían sido tratados en los servicios de cirugía, UCI, reanimación quirúrgica, urgencias, geriatría o en un centro de atención primaria. Los pacientes eran tres mujeres y cuatro varones de edades entre 45 a 86 años.
Tenemos que destacar que todos nosotros vivimos en armonía con nuestras bacterias. Bacterias en nuestra boca, en la piel y en nuestro intestino. La bacteria de nuestro estudio, E. coli es un miembro común de nuestra microbiota intestinal y forma parte de las llamadas “bacterias comensales”, que viven con nosotros en una relación respetuosa y beneficiosa. Cuando alguna de estas bacterias cambia de posición y llega a una localización inadecuada provoca una infección, llamándose “patógeno oportunista”. Esto ocurre, por ejemplo, cuando E. coli llega al aparato urinario y causa una cistitis. En el caso de E. coli, puede causar un amplio rango de enfermedades, desde diarreas de distinta gravedad a patologías extraintestinales. Éstas pueden provocar infecciones en sistema nervioso central (provocando meningitis), infección sistémica (bacteriemia) o vía urinaria (infecciones de orina).
Además de los determinantes de virulencia, necesarios para colonizar nuevos nichos, las bacterias pueden adaptarse para su supervivencia adquiriendo genes de resistencia a antibióticos, o a otros compuestos químicos, como metales pesados o desinfectantes. ¿Qué estrategias siguen las bacterias para ser inmunes a los ataques “químicos” que les proferimos? De una manera sencilla, y asemejándolo a un sistema llave-cerradura. La llave, los antibióticos son moléculas que para realizar su función de bloqueo deben unirse a su cerradura, la diana. Estas moléculas diana son vitales para el funcionamiento y supervivencia de la bacteria, son puntos vitales. Por este motivo, la unión del antibiótico a su diana provoca la muerte bacteriana. Pero las bacterias han encontrado la manera de bloquear los antibióticos, y han desarrollado mecanismos sofisticados para evadir su acción. Por ejemplo, pueden reducir la permeabilidad de su pared bacteriana, e impedir que el antibiótico entre en la célula; o una vez que ha entrado, pueden expulsarlo mediante bombas de expulsión activa; pueden hiperproducir o modificar la diana, de manera que el antibiótico no pueda destruirlo; o incluso inactivar o modificar el antibiótico. En todos estos casos, el antibiótico será inservible para eliminar la bacteria y ésta se habrá convertido en resistente. (Figura 4).
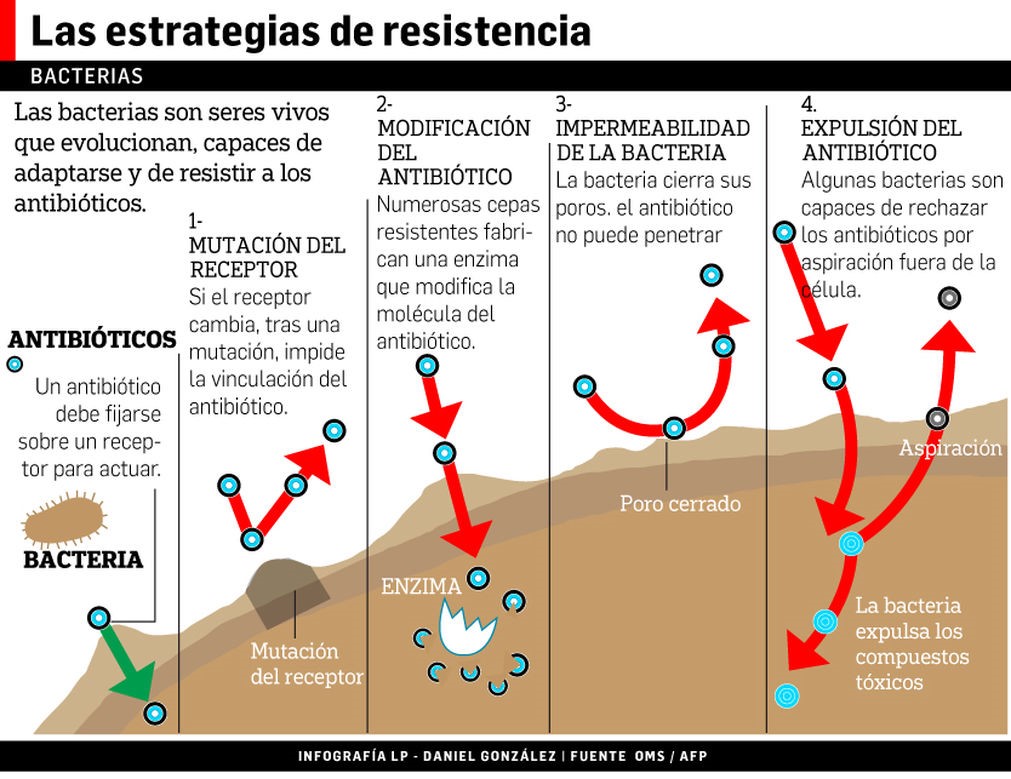
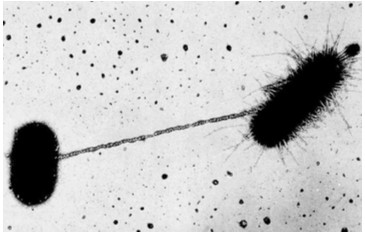
Pero además, las bacterias en su afán de supervivencia comparten los genes con otras bacterias mediante los Elementos Genéticos Móviles, transmitiendo su resistencia. Existen diversos tipos de Elementos Genéticos Móviles, clasificados por su manera de transferirse. Entre ellos destacan los plásmidos, que portan los genes de resistencia a antibióticos, a metales pesados, desinfectantes y virulencia, y son intercambiados en un proceso de conjugación. Este intercambio de material genético enriquece a la población bacteriana y le provee de ventajas adaptativas para sobrevivir en ambientes hostiles. (Figura 5).
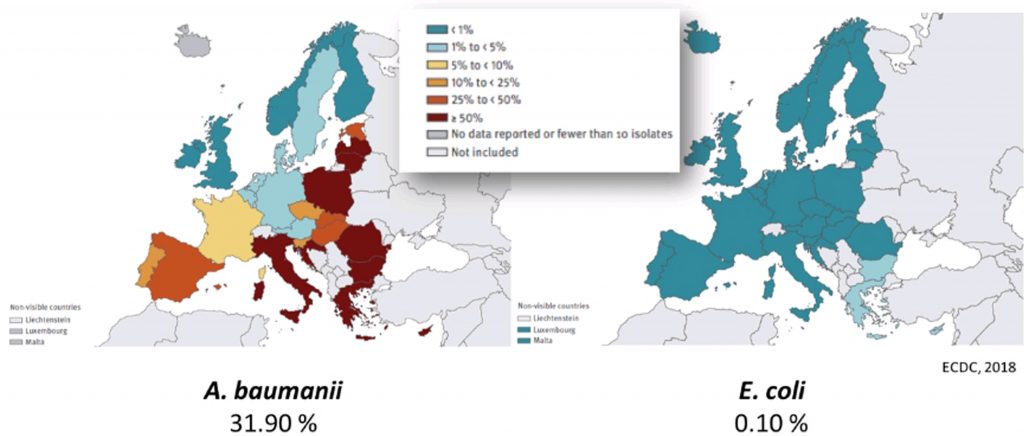
Lo que realmente nos preocupaba de lo que estaba ocurriendo en el Hospital Central de Asturias era la resistencia a antibióticos carbapenémicos. Éstos son antibióticos de amplio espectro, es decir, son eficaces frente a un gran número de tipos bacterianos, y por tanto, son una de las “balas” más potentes que tenemos frente a las infecciones complicadas. Quizás les suenen ertapenem, imipenem o meropenem, que son utilizados para el tratamiento de infecciones graves en los hospitales.. La resistencia a este tipo de antibióticos a nivel europeo varía en función de la bacteria y el país que estemos analizando. Por ejemplo, para Acinetobacter baumanii (un patógeno común en hospitales)latasa media de resistencia carbapenémicos es superior al 30%. Hay discrepancias entre el sur de Europa, donde habitualmente las tasas son más altas, y el norte de Europa, con tasas menores al 1% de incidencia. Sin embargo, la tasa de resistencia a antibióticos carbapenémicos en E. coli, el objeto de nuestro trabajo, es de 0.10% a nivel Europeo, siendo menor al 1% en casi todos los países analizados. Por esta razón, encontrar siete casos de pacientes infectados por E.coli resistente a carbapenémicos en un único hospital resultaba especialmente alarmante. (Figura 6).
Ante esta situación nuestras preguntas se centraron en: ¿estamos ante un brote epidémico? ¿son situaciones esporádicas en el hospital? y ¿qué riesgo de diseminación de esta bacteria tenemos?
Para responder a estas preguntas analizamos el genoma bacteriano mediante técnicas de secuenciación masiva. Estas técnicas son como una lupa gigante, que nos permiten leer cada uno de los componentes del genoma, de la misma manera que se empleó para el Proyecto Genoma Humano. Somos capaces de generar millones de datos en muy pocas horas, que después debemos analizar mediante técnicas bioinformáticas, combinando conocimientos biológicos, estadísticos, matemáticos y programación avanzada. Nuestro primer paso fue comparar los genomas de nuestras bacterias frente a otros genomas que están disponibles en bases de datos internacionales, realizando un análisis filogenético. Observamos que los genomas 1, 2, 3 y 4 tenían mayor relación con genomas aislados en otras zonas del mundo y de otro tipo de muestras, no necesariamente clínicas, que entre ellos mismos. Esto nos hacía descartar un posible brote epidémico localizado entre los años 2012 a 2014. Sin embargo, los genomas 5, 6 y 7 estaban mucho más relacionados entre ellos, indicando que éstos, aislados en todos los casos en el año 2015, sí que podrían ser un brote epidémico. Aunque observábamos dos situaciones distintas, todas estas bacterias tenían en común un gen codificante para una enzima, OXA-48, que degrada el antibiótico carbapenémico.
Nuestras preguntas no acababan aquí. Necesitábamos saber cuál era el riesgo de diseminación de este tipo de resistencia. Para ello centramos el análisis bioinformático en los Elementos Genéticos Móviles. Nuevamente, comparamos los plásmidos obtenidos de los genomas 1, 2, 3 y 4, frente a plásmidos procedentes de otros lugares del mundo. Todos ellos llevan únicamente la resistencia a los antibióticos carbapenémicos, y el resto del plásmido es la maquinaria necesaria para auto-transferirse de una manera rápida y efectiva entre la población bacteriana. La primera vez que se describió este plásmido fue en el año 2012 en el Sultanato de Omán, a partir de un paciente infectado por el patógeno Klebsiella pneumoniae. Desde ese momento, se ha detectado este elemento genético móvil en distintos tipos de bacterias patógenas, prácticamente en todos los países del mundo y no sólo en muestras clínicas, sino también de animales de compañía o de consumo. No sabemos muy bien cómo llegó hasta el Hospital Universitario Central de Asturias, ya que recordemos que las bacterias son “viajeras”, pero de lo que sí que estamos seguros, es de que este plásmido se encontraba circulando en este hospital.
Finalmente, la última parte del estudio se centró en el análisis in vivo de estos genomas, para poder analizar su virulencia en un modelo animal, puesto a punto en la U. de Santiago de Compostela. Se utilizaron diez ratones hembra por grupo, inoculando una preparación bacteriana subcutánea. Se midió letalidad entre las 24 h y 7 días post-inyección, examinándose diariamente los signos de necrosis. La letalidad fue menor de un 10%, ya que sólo tres cepas causaron fallecimiento en uno de los diez ratones inoculados. Todas ellas mostraron una respuesta local inflamatoria con una necrosis subcutánea extensa en los ratones que sobrevivieron. Éste ha sido el primer estudio internacional en demostrar la virulencia en vivo de este tipo de cepas. Los resultados eran consistentes con la patología observada en los pacientes donde ninguno cursó sepsis.
Por lo tanto, este trabajo nos permitió responder a las preguntas que nos planteamos. Pudimos diferenciar mediante técnicas de secuenciación masiva, aquellos casos en los que la resistencia venia dada por la dispersión de un Elemento Genético Móvil circulante entre poblaciones bacterianas, a un caso concreto de brote epidémico. Actualmente, mediante dos proyectos financiados por el Instituto de Salud Carlos III, continuamos analizando Enterobacterias productoras de carbapenemasas en hospitales de la cornisa cantábrica, como son los hospitales de Donosti, Vitoria, Cruces, Valdecilla o el Hospital Universitario Central de Asturias, entre otros. (Figura 7).

Son muchos los problemas que tenemos que abordar de manera continua y coordinada, integrando a todos los profesionales sanitarios, incluidos los responsables de sanidad animal, antes de que necesitemos más tiempo, más esfuerzo y más recursos para revertir la situación. Los investigadores científicos trabajamos con pasión y dedicación para ayudar a la sociedad, desde la investigación más básica, hasta la aplicación más clínica o tecnológica. Este premio no ha hecho más que reforzar la filosofía con la que nuestro grupo trabaja: la importancia del trabajo en equipo y del enfoque multidisciplinar, frente a la endogamia científica y al trabajo unidisciplinar. Debemos unir nuestras fuerzas para mitigar y reducir el efecto de la resistencia a antibióticos, no sólo en nuestra salud, sino también sobre animales y medio ambiente, con el fin de preservar la eficacia de los antibióticos existentes. (Figura 8).
Y es que, los humanos en realidad no somos tan brillantes. Pensamos en muchas cosas a lo largo del día, pero las bacterias sólo piensan en una: sobrevivir (Gary French, Hospital St. Thomas, Londres Sunday Times. 2000).

María de Toro & Javier Fernández (Centro de Investigación Biomédica de La Rioja & Hospital Universitario Central de Asturias).




